Assemblages Macro-Moléculaires
Ce cours a comme objectif la présentation des aspects structuraux spécifiques aux assemblages macromoléculaires et aux demarches expérimentales pour les caracteriser. Le cours décrit des aspects pertinants de la structure des assemblages tels que la morphoologie, la stabilité relative des assemblages, les caracteristiques des surfaces d'interaction entre monomères, les relations de symmétrie, etc. Au delà d'un rappel sur les démarches de biologie structurale sous-jacentes, le cours fait la présentation de différents assemblages de référence, qui permettent d'illustrer la diversité des interactions et l'illustration de caractéristiques communement trouvées dans ces structures. Des interventions par des chercheurs et par les étudiants suivant le cours permettent d'aborder différentes thématiques structurales.
- Introduction
- Taille des Protein Protéines: Oligomerisation; Symétrie; Propriétés de Surface
- Méthodes d'Analyse Structurale; Comparaison Cristallographie+RMN+Microscope Electronique; Notion de Tomographie; Multi-résolution
- Protéines Désordonnées (SL)
- Examples d'Assemblages: Assemblages Viraux et leurs Relations Structure-Fonction
- Examples d'Assemblages: Clatherine; Dynamine; Cytoskelette; Moteurs Moléculaires; Etc
- Présentations d'Articles par les Etudiants
- TP: Introduction pratique à la Cryo-microscopie (Préparation de Grilles; 1ère analyse d'Images)
- Enseignant: Rhian JONES
- Enseignant: Pedro MALDONADO COUTINHO
- Enseignant: Denis PTCHELKINE
- Enseignant: Latifa ELANTAK
- Enseignant: Marie line GARRON
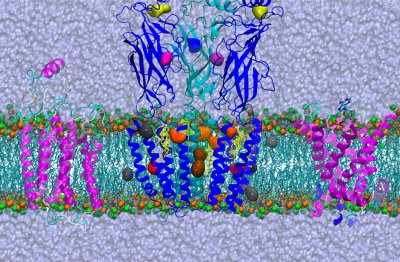
Modélisation moléculaire - JP Duneau (6H cours-TD + 6H TP)
-Mécanique moléculaire et champs de forces
-Modélisation basée sur les connaissances
-Modélisations ab initio
Diffusion moléculaire et modélisation - A. Sergé (6H cours-TD)
Docking moléculaire et drug design - S. Betzi (6H cours-TD + 6H TP)
- Enseignant: Stephane BETZI
- Enseignant: Jean pierre DUNEAU
- Enseignant: Arnauld SERGE
- Enseignant: Carole BAFFERT
- Enseignant: Myriam BRUGNA
- Enseignant: Sebastien DEMENTIN
- Enseignant: Jean pierre DUNEAU
- Enseignant: Emilien ETIENNE
- Enseignant: Stephane GRIMALDI
- Enseignant: Bruno GUIGLIARELLI
- Enseignant: Marlene MARTINHO
- Enseignant: Elisabetta MILEO
- Enseignant: Eric PILET
- Enseignant: James STURGIS
Dynamique des réseaux d'intéractions moléculaires
- Approches biochimiques
- Co-évolutions et approches théoriques
Dynamique enzymatique
- Réaction à 2 substrats (enz. Formelle)
- Etat prestationnaire (Stop flow et temp. Jump, relaxation cinétique)
- Effets isotopiques (y compris mécanismes)
- Régulation
Dynamique et thermodynamique
- FRAP, FCS
- EPR et marquage de spin
- Protéines Intrinséquement Désordonnées
Simulations
- Relation structure-dynamique fonction
- Champs de forces
- Principes et mise en œuvre
- TP de simulation de dynamique moléculaire
- Enseignant: Gilles BREUZARD
- Enseignant: Myriam BRUGNA
- Enseignant: Jean pierre DUNEAU
- Enseignant: Corinne KREUZER
- Enseignant: Marlene MARTINHO
- Enseignant: Eric PILET
- Enseignant: James STURGIS
- Enseignant: Frederic BIASO
- Enseignant: Benedicte BURLAT
- Enseignant: Elsa GARCIN
- Enseignant: Marianne ILBERT
- Enseignant: Sonia LONGHI
- Enseignant: James STURGIS